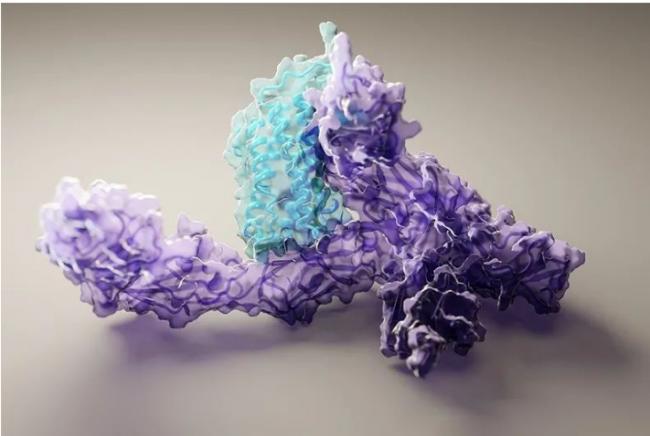
理解蛋白質的結構,有助於確定蛋白質的功能,了解各種突變的作用。截至目前,約有10萬個蛋白質的結構已經用實驗方法得到了解析,但這在已經測序的數10億計的蛋白質中只佔了很小一部分。
幾十年來,研究人員一直使用 X射線晶體學和冷凍電子顯微鏡等實驗技術來確定蛋白質結構。但是這樣的方法既費時又費錢,而且一些蛋白質不適合這樣的分析。
在過去50多年的時間裏,研究人員一直嘗試根據蛋白質的氨基酸序列預測其折迭而成的三維結構。然而,當前使用的計算方法準確度有限,實驗方法對人力和時間的要求也非常高。
2021年7月16日,谷歌公司旗下 DeepMind團隊在 Nature發表了題為:Highly accurate protein structure prediction with AlphaFold的研究論文。
該論文描述了神經網絡AlphaFold2,AlphaFold2能以前所未有的準確度根據蛋白質的氨基酸序列預測其三維結構。
同時,DeepMind團隊在論文中公佈了AlphaFold2的開原始碼,並發表了系統的完整方法論,詳盡細緻說明AlphaFold是如何做到精確預測蛋白質3D結構的。

Demis Hassabis等人描述了AlphaFold2,這是一個基於神經網絡的新模型,其預測的蛋白質結構能達到原子水平的準確度。作者們在2020年5-7月舉辦的第14屆「蛋白質結構預測關鍵評估」(CASP14)大賽中驗證了這種方法。
CASP14要求參賽團隊根據蛋白質的氨基酸序列解析它們的結構。比賽用的蛋白質會先用實驗方法解析出來,但具體結果不會公開。比賽中,AlphaFold2預測的大部分結構達到了空前的準確度,不僅與實驗方法不相上下,還遠超解析新蛋白質結構的其他方法。
將實驗方法得到的蛋白質結構迭加在AlphaFold2的結構上,組成蛋白質主鏈骨架的迭加原子之間的距離中位數(95%的覆蓋率)為0.96埃(0.096納米)。成績排第二的方法只能達到2.8埃的準確度。
AlphaFold2的神經網絡能在幾分鐘內預測出一個典型蛋白質的結構,還能預測較大蛋白質(比如一個含有2180個氨基酸、無同源結構的蛋白質)的結構。該模型能根據每個氨基酸對其預測可靠性進行精確預估,方便研究人員使用其預測結果。
論文作者認為,這一精準的預測算法可以讓蛋白質結構解析技術跟上基因組革命的發展步伐。

Demis Hassabis
更重要的是,該論文的通訊作者、DeepMind創始人Demis Hassabis發佈了一項聲明:
去年在CASP14大會上我們揭曉了一個可以將蛋白質3D結構預測精確到原子水平的全新AlphaFold系統,此後我們承諾會分享我們的方法,並為科學共同體提供廣泛、免費的獲取途徑。今天我們邁出了承諾的第一步,在 Nature期刊上分享AlphaFold的開原始碼,並發表了系統的完整方法論,詳盡細緻說明AlphaFold是如何做到精確預測蛋白質3D結構的。作為一家致力於推動科學進步的公司,我們期待看到我們的方法將為科學界啟發出什麼其他新的研究方法,也期待很快能和大家分享更多我們的新進展。」
值得一提的是,就在同一天,Science期刊發表了來自華盛頓大學 David Baker團隊的蛋白質結構預測論文,開發了名為 RoseTTAFold的蛋白質結構預測系統。

David Baker表示,2020年 DeepMind在CASP14大會上的表現,給業界帶來極大震撼,AlphaFold系統對蛋白結構的預測如此精準,讓學術界產生了許多悲觀情緒,他自己甚至一度覺得要失業了。
但他很快調整心態,決定挑戰AlphaFold系統,並很快開發出了與AlphaFold系統媲美的 RoseTTAFold系統,而且該系統不僅可以預測蛋白質結構,還能預測蛋白複合物結構。
6月中旬,David Baker將論文在預印本上線,三天後 DeepMind創始人Demis Hassabis表示 AlphaFold2的詳細信息將登錄 Nature並免費開源。David Baker也將 RoseTTAFold系統免費開源,供所有人下載使用。

現在,所有人都可以免費獲得 RoseTTaFold和 AlphaFold2的代碼,研究人員將能夠在這兩項進展的基礎上再接再厲,蛋白質結構解析將在人工智能的幫助下走向大眾。